Nat Commun:机器学习分类器CUPiD:基于cfDNA甲基化模式预测原发灶不明癌症的起源组织
2024-05-13 测序中国 测序中国 发表于上海
文章介绍了一个高度准确的机器学习分类器CUPiD,可基于cfDNA甲基化模式准确预测29种肿瘤类型的TOO。
导读
原发灶不明癌症(CUP)是一种经病理学检查确诊为转移性肿瘤,但经过详尽的临床检测和评估手段仍无法明确其原发灶的恶性肿瘤,病程短、进展快。由于生物异质性较高和对标准化疗反应不佳,目前CUP的诊断和治疗是一个日益重要但尚未解决的临床难题。近年来,生物标志物驱动的精确肿瘤学及免疫疗法的出现改进了肿瘤的标准治疗方案,并提高了许多肿瘤类型的总生存率,但仅有少数治疗方法被批准用于CUP患者,有大多数CUP患者无法从中获益。
预测CUP的起源组织(TOO)的分子表征方法被认为可更好地实现分层治疗,但基于TOO预测的治疗是否能改善结果仍存在争议,其易受到预测肿瘤类型的异质性、多样性以及次优治疗的阻碍。此外,对CUP患者进行分子分析的另一个重大挑战是缺乏用于分析的高质量肿瘤组织。已有研究提出了一种稳健、敏感的全基因组循环游离DNA(cfDNA)甲基化分析流程(T7-MBD-seq),可实现对患者的灵敏检测、癌症发生发展监测及分子亚型分层。
近日,英国曼彻斯特大学的科研人员在Nature Communications发表了题为“A cfDNA methylation-based tissue-of-origin classifier for cancers ofunknown primary”的文章,介绍了一个高度准确的机器学习分类器CUPiD,可基于cfDNA甲基化模式准确预测29种肿瘤类型的TOO。研究团队使用来自13种癌症类型患者以及27名非癌症对照的143个cfDNA样本对CUPiD进行验证,总体灵敏度为84.6%,TOO准确度为96.8%;在另一组队列对78.0%CUP患者(共41例)进行CUPiD预测,其中88.5%的预测与临床上诊断结果一致。因此,将CUPiD与cfDNA突变数据相结合可促进对肿瘤患者的诊断和治疗分层,并改善CUP群体的预后。

文章发表在Nature Communications
主要研究内容
基于cfDNA甲基化数据开发TOO分类器CUPiD
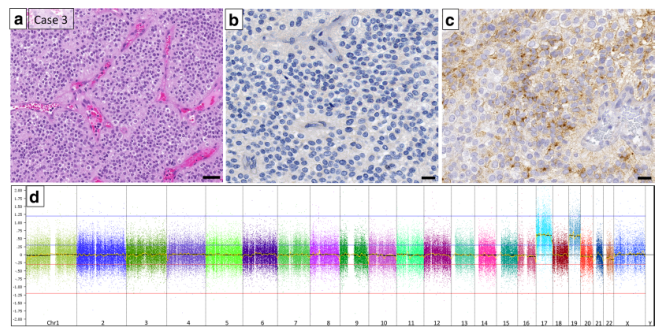
为构建适用于cfDNA样本的稳健多类别TOO分类器,首先应解决cfDNA中循环肿瘤DNA(ctDNA)的高变异性(肿瘤分数, TF)问题,其会导致非癌cfDNA稀释主要的肿瘤特异性信号。为克服这一难题,研究团队通过一种生物信息学方法来模拟具有不同TF的cfDNA样本。使用公开可用的肿瘤组织DNA甲基化数据(主要来自TCGA),涉及29种肿瘤类型(9,017个肿瘤),将甲基化β值转换为估计的T7-MBD-seq计数;并将计数与先前测序的非癌症个体(非癌症对照,NCC)cfDNA样本混合,创建模拟混合物。最终,共创建276,108个混合物,包含30个类型。
随后,研究团队使用差异甲基化区域(DMR)计算肿瘤特异性基因组区域,并对30个类别进行比较,选择了差异最大的250个DMRs;对这些DMR进行降维处理并应用于9,017个肿瘤样本。基于上述数据,研究团队建立了一个名为CUPiD的集成分类器,由100个独立的梯度增强树子分类器组成,其中每个子分类器均在肿瘤患者队列和NCC混合模拟数据上进行了训练。经验证,CUPiD的平均多类别受试者操作曲线下面积(AUROC)为0.984。
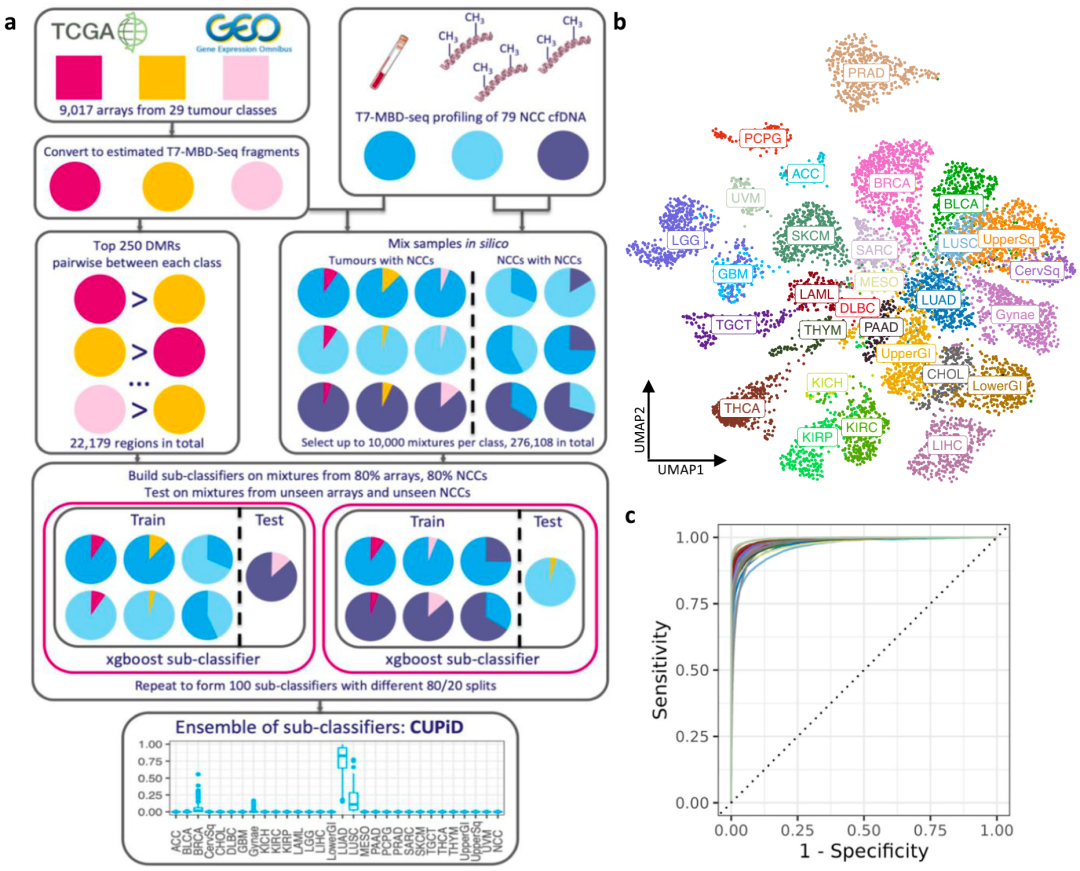
图1. 基于cfDNA甲基化数据的组织来源分类器的构建
CUPiD在多种癌症类型中的性能
研究团队在170个cfDNA样本的独立测试队列中测试了CUPiD,其中包括来自13种不同肿瘤类型的143个癌症患者样本和27个使用T7-MBD-seq分析的NCC样本。在癌症cfDNA样本中,CUPiD预测了121例患者的正确肿瘤类型,总体灵敏度为84.6%,4例患者预测不准确(2.8%),另有18例患者未得到分类(12.6%);27个NCC样本中均未被预测为肿瘤类别。此外,在具有肿瘤预测的125个样本中,CUPiD准确预测了121个病例,TOO准确度为96.8%。与正在开发的用于早期癌症检测的其他cfDNA TOO分类器相比,CUPiD更具优势,准确率更高。上述结果表明,基于甲基化图谱能够检测肿瘤信号并可准确预测TOO。
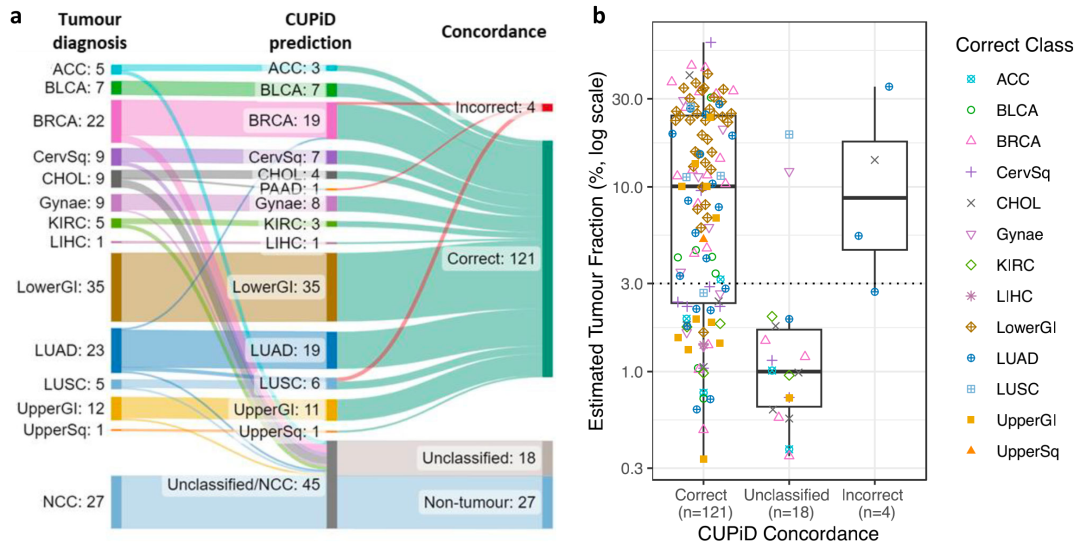
图2. CUPiD在已知肿瘤类型患者队列中的性能验证
基于CUP队列的cfDNA来确定TOO
在一项41例CUP患者的初步研究中,研究团队评估了将cfDNA甲基化和突变分析与TOO预测相结合的可行性。往期临床数据(包括临床病史、病理学等)显示,41例患者中有15例(36.6%)例在之后被确诊为原发性肿瘤并接受治疗(“临床消退”);18例进行了“疑似肿瘤诊断”;8例未进行潜在的原发性肿瘤诊断(“无临床怀疑”)。
研究团队首先对上述整个队列进行了成功的cfDNA甲基化分析,预估的ichorCNA肿瘤分数TF为0-53.4%,其与通过突变分析计算的中位VAF相关良好;进一步,使用CUPiD预测该队列中的TOO。结果显示,CUPiD在32例患者(78.0%),中产生了肿瘤预测,对9例患者(22.0%)进行了未分类预测。在32个肿瘤预测中,26个患者(81.3%)为5大最常见的预测癌症类别,即肝胆胰癌(7例)、女性生殖道癌(6例)、上/下消化道癌症(4例)、肺癌(5例)和泌尿系癌症(4例),这与CUP患者尸检中发现的原发性肿瘤类型一致。
有趣的是,与CUP标准治疗方案(化疗)相比,所有CUPiD预测的肿瘤类型都具有其他不同的治疗策略,且几乎所有预测都指向需要考虑将免疫治疗或靶向治疗作为一线或二线治疗选择,这体现了CUPiD的临床应用潜力。
在33例“临床消退”或疑似诊断的患者中,26例具有CUPiD肿瘤类型预测,其中23例(88.5%)与确诊的原发性肿瘤类型或疑似诊断类型一致,3个CUPiD预测与临床数据不一致,为错误分类。
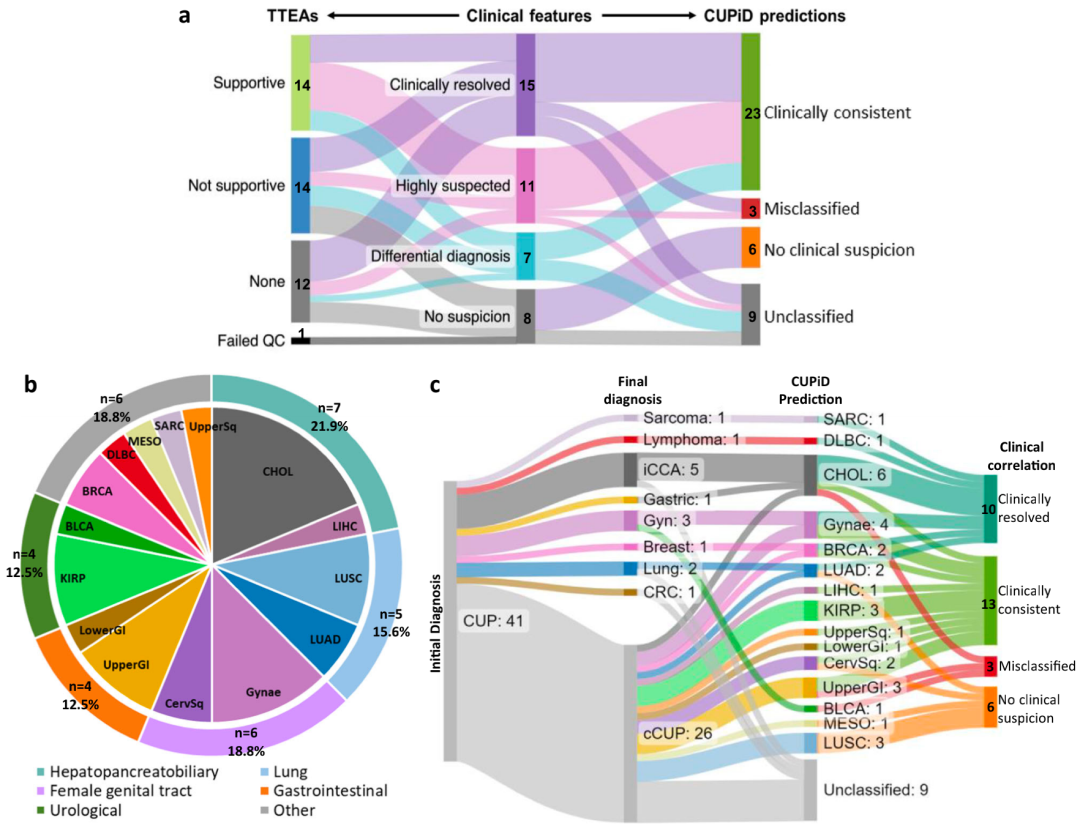
图3. CUPiD在41例CUP患者cfDNA检测中的应用
CUPiD的潜在临床效用
15例原发性肿瘤“临床消退”患者经历了长期的诊断不确定性,大多数患者在确诊原发性肿瘤之前接受了次优经验性化疗。该队列患者的中位诊断时间为7.1个月,6例患者进行了侵入性重复活检才最终确诊。当有足够的IF时,在疑似癌症诊断甚至组织活检前,使用基于液体活检的TOO分类器进行预测,如CUPiD,可显著加速大部分患者的确诊,并可能避免重复侵入性活检。此外,珍贵的肿瘤组织材料可以保留用于潜在的组织生物标志物检测,大多数靶向治疗和一些免疫治疗开始前都需要这些材料。目前,CUPiD的实验周转时间为3周,后续有望通过优化检测程序进一步缩短周转时间。
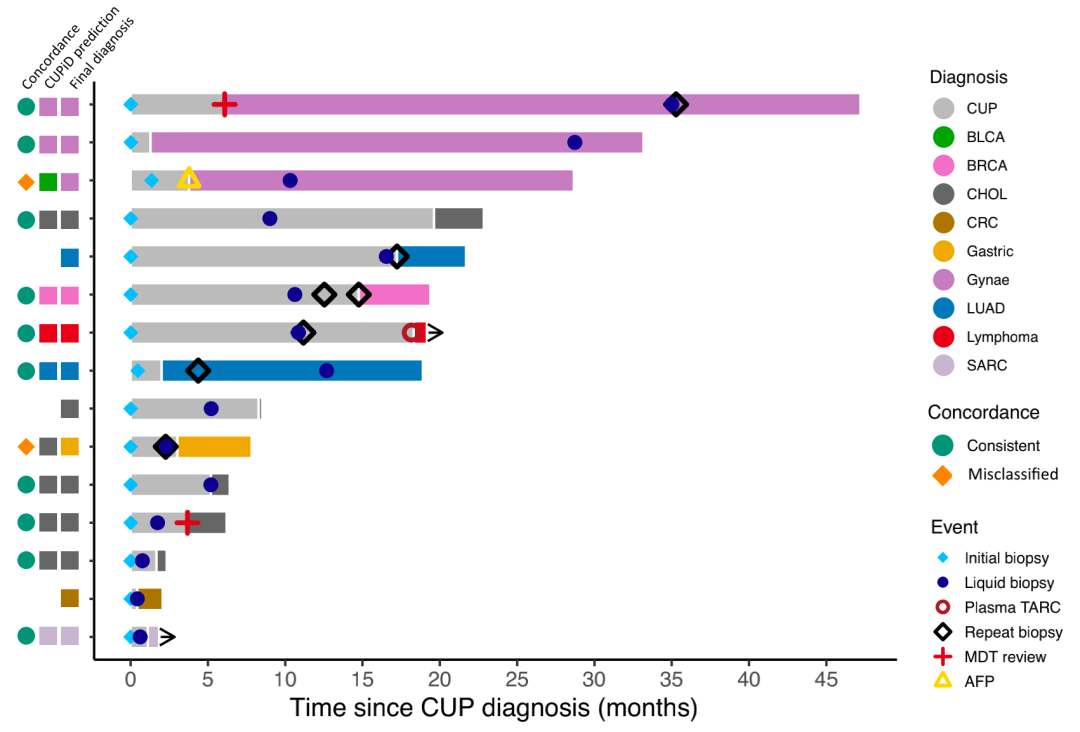
图4. 15例原发性肿瘤“临床消退”患者的治疗持续时间
结语
综上所述,研究团队开发了CUPiD,这是一种基于液体活检的TOO分类器,在已知肿瘤类型中具有较高灵敏度和准确性,并对cfDNA含量足够TF的CUP患者具有与临床一致的预测。特别地,由于cfDNA突变和甲基化分析可从同一次抽血样本中进行,该方法能够识别潜在可操作的改变,进行准确TOO预测,帮助医生对患者进行分层诊断和治疗。此外,与标准治疗方案相比,CUPiD成功对78%CUP患者的肿瘤类型进行预测,扩大了可从肿瘤特异性治疗策略中获益的CUP患者范围。
据悉,接下来研究团队将在在更大的已知肿瘤cfDNA样本队列和具有统计学效力的前瞻性CUP临床试验中进一步验证CUPiD。
参考原文:
Conway, AM., Pearce, S.P., Clipson, A. et al. A cfDNA methylation-based tissue-of-origin classifier for cancers of unknown primary. Nat Commun 15, 3292 (2024). https://doi.org/10.1038/s41467-024-47195-7.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言















#甲基化# #cfDNA# #原发灶不明癌症#
10